오늘은 BERT의 친구인 BioBERT논문을 보려고 합니다. 이 논문은 2019년에 Bioinformatics라는 저널에 올라온 자랑스러운 한국인이 작성한 논문입니다. 실제로 인용 수도 966회로 나름 많습니다. 그럼 이제 살펴볼까요?
Biomedical document의 수가 점점 많이 증가함으로 인해서 biomedical text mining이 점점 중요해지고 있습니다. NLP기술의 진보로 인해 biomedical문헌에서 중요한 정보를 추출하는 것이 연구자들 사이에서 인기를 얻었고, 딥러닝은 효과적인 biomedical text mining 모델의 개발을 촉진시켰습니다. 그러나 NLP의 발전과는 다르게 이러한 기술을 biomedical text mining에 직접 적용하면 제대로 된 결과물을 얻기 힘듭니다. 왜냐하면 단어 분포가 대부분의 모델의 경우 general domain corpora인데 반해 여기서는 biomedical corpora라는 특수한 단어 분포를 가지기 때문입니다. 그래서 이 논문에서는 pre-trained language model인 BERT가 biomedical corpora에 잘 적용될 수 있는지 알아봅니다.
Introduction
엄청난 수의 biomedical관련 report들이 계속 쌓여가고 있는 중입니다. PubMed라는 곳 한곳만 봐도 2019년 1월 기준 29백만개의 article들이 쌓여있다고 합니다. 그런데 앞으로 출판되는 report들은 점점 더 많아지기 때문에 biomedical관련해서 중요한 정보를 추출할 수 있는 text mining을 위한 수요가 증가하고 있습니다. 다행히도 최근 biomedical text mining 모델은 NLP에서 사용되는 딥러닝 기술의 진보에 의해서 발전되고 있습니다. 예를 들어서 지난 몇 년 동안 LSTM이나 CRF와 같은 기술들은 biomedical 이름 인식(NER)에 대하여 굉장한 성능을 보였습니다. 이외에도 biomedical text mining을 위한 다른 딥러닝 기반 모델들도 관계 추출(RE)과 질의 응답(QA)관련해서 진보된 성능을 보여줬습니다.
그런데 사실 state-of-the-art NLP 모델을 biomedical text mining에 직접 적용하는 것은 한계가 존재합니다. 첫 번째 이유로 최근 소개되는 word representation 모델들인 Word2Vec, ELMo, BERT 등은 위키피디아문서와 같은 일반적인 데이터 셋 도메인에서 학습되고 테스트되기 때문입니다. 이렇게 되면 biomedical text들이 포함된 데이터 셋에 대해서 성능 측정을 하기 어렵습니다. 또한 일반적인 단어 분포와 biomedical text의 분포가 서로 다릅니다. 이러한 이유때문에 최근 biomedical text mining 모델들은 word representation이 biomedical에 적용된 버전에 크게 의존하게 됩니다.
이런 연구결과들로 인해서 이 논문에서는 word representation 모델의 state-of-the-art 모델인 BERT(이 당시에는 채고 잉기모델 이었습니다)가 효과적인 biomedical text mining 문제를 풀기 위해 biomedical corpora에 대한 학습이 필요하다고 가설을 세웠습니다. 또한 BERT는 다양한 NLP 문제에 대해서 거의 동일한 아키텍처로 강력한 성능을 입증했습니다. 그렇기에 biomedical 도메인에 BERT를 적용하면 다양한 biomedical NLP 연구에 잠재적으로 도움이 될 수 있을 것입니다.
Approach
이제부터는 이 논문에서 소개하는 BIoBERT에 대해서 알아보겠어요. BioBERT는 biomedical 도메인으로 pre-trained된 language representation 모델입니다. BioBERT의 pre-training과 fine-tuning의 전체 과정은 다음 그림에서 볼 수 있습니다. 첫 번째로, 이 논문에서는 BERT로부터 추출된 weight를 사용해서 BioBERT를 초기화 합니다. 즉, 일반 도메인 corpora인 영문 위키피디아와 BooksCorpus로부터 pre-trained된 BERT의 weight를 사용한다는 의미입니다. 그러고나서 그림에서도 볼 수 있듯이 BioBERT는 biomedical 도메인 corpora인 PubMed와 PMC를 통해서 pre-trained됩니다. 다음으로 이렇게 pre-trained된 모델을 통해서 biomedical text mining에 대해 좋은 결과를 낸다는 것을 보여줍니다. 즉, BioBERT는 3가지 유명한 biomedical text mining 문제인 NER, RE, QA에 대해서 fine-tuned되고 평가됩니다. 이를 위해 이 논문에서는 일반적인 도메인 corpora와 biomedical corpora를 다양한 조합과 크기로 pre-training 전략들을 테스트하고 각 corpus가 pre-training에 미치는 영향을 분석합니다(biomedical text만으로 pre-trained 모델을 만드는게 아니라 일반 도메인까지 사용합니다).
실제로 biomedical corpora에 대한 pre-training BERT는 기존 state-of-the-art보다 성능이 더 좋다고 합니다. 또한 이전 biomedical text mining 모델 연구들은 NER이나 QA와 같이 한가지 문제에만 초점을 맞춰서 진행한 반면에 BioBERT는 약간의 아키텍처 수정만으로 다양한 biomedical text mining 문제들에 대해서 state-of-the-art 성능을 보여줬습니다.

Materials and methods
BioBERT는 기본적으로 BERT와 같은 구조를 가집니다. 그렇기에 BERT에 대해서 간단하게 설명한 후 BioBERT의 pre-training과 fine-tuning에 대해서 자세히 알아보겠습니다.
BERT
일단 이전의 Word2Vec과 같은 word representation 모델들은 context에 독립적으로 word representation을 했는데(그니까 문장의 문맥과는 상관없이 단어를 표현하는 것을 의미해요. 먹는 배, 타는 배, 뚱뚱한 배 이런 배를 같은 벡터로 매핑하는 거예요), 최근에는 context와 dependent하게 word representation을 합니다.
BERT는 대표적인 contextualized word representation 모델입니다. 어떻게 그러면 이런 context를 잘 학습할 수 있었냐 하면 다음과 같습니다. 보통 다른 자연어 모델들은 그 모델 자체의 특성으로 인해 미래의 단어를 볼 수 없다는 한계가 존재합니다. 즉, 왼쪽에서 오른쪽으로 혹은 오른쪽에서 왼쪽으로 가는 단방향 모델입니다. 하지만 BERT는 masked language model을 사용하고, transformer 구조의 encoder부분만을 사용해서 bidirectional representation을 사용할 수 있습니다. BERT의 저자는 이렇게 bidirectional representation의 정보를 통합하는 것이 자연어로 word representing하는데 중요하다고 합니다. 이 논문의 저자도 biomedical term 사이의 복잡한 관계가 종종 biomedical corpus에 존재하기 때문에 이러한 bidirectional representation이 biomedical text mining에서 중요할 것이라고 가정합니다.
Pre-training BioBERT
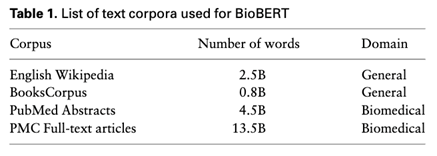
앞에서 설명한 BERT와는 다르게 BioBERT는 biomedical 도메인에서의 word representation을 지향하는데, biomedical 도메인에는 일반 도메인과는 다르게 다양한 고유명사 및 용어가 포함되어 있습니다. 따라서 범용 언어 이해를 목적으로 하는 NLP 모델은 biomedical text mining 문제에서 좋지못한 결과를 냅니다. 그래서 이 논문에서는 BioBERT를 PubMed abstracts(PudMed)와 PudMed Central full-text article(PMC)에 대해서 pre-train 시킵니다. BioBERT를 pre-training할 때 사용하는 text corpora는 아래 Table 1에 나타나 있습니다. 그리고 text corpora의 조합에 대한 테스트 정보는 아래 Table 2에 나타나 있습니다.

이 논문에서는 pre-trained BERT로 BioBERT를 초기화하고, 이를 통해 BioBERT를 biomedical corpora를 포함하는 pre-training corpora language representation 모델로 정의합니다(예를 들어 BioBERT + PubMed).
Fine-tuning BioBERT
BioBERT의 아키텍처를 조금만 변경해도 다양한 downstream text mining 문제에 BioBERT를 적용할 수 있습니다. 이 논문에서는 3가지의 representation biomedical text mining 문제에 fine-tune BioBERT를 적용합니다(NER, RE, QA).
먼저 NER에 대해서 봅시다. NER은 Named entity recognition의 약자로 biomedical text mining 에서 가장 기초적인 문제 중 하나입니다. NER은 수많은 biomedical corpus에서 domain-specific한 고유 명사를 인식하는 것입니다. 이 문제를 위해 BERT는 last layer로부터의 representations를 기반으로 single output layer를 사용하여 확률을 계산합니다.
다음으로 RE인 Relation extraction 입니다. 이 문제는 biomedical corpus에서 명명된 entity들의 관계를 분류하는 문제입니다. 관계를 분류하기 위해 [CLS]토큰을 사용하는 BERT의 sentence classifier를 활용했습니다. Sentence classification은 BERT의 [CLS] 토큰 representation을 기반으로 single output layer를 사용해서 적용됩니다.
QA는 Question answering 입니다. 이는 관련된 구절이 주어지면 자연어로 질문에 대한 답변을 하는 문제입니다.
이렇게 마지막으로 fine-tuning에 대해서도 알아보았습니다.
Conclusion
이 포스터에서는 BioBERT를 소개했습니다. BERT와 비슷하게 BioBERT는 pre-trained language representation model 이며, 특히 biomedical text mining 쪽에 특화되어 있습니다. 위에서 살펴봤듯이 biomedical corpora에 대한 pre-training BERT 는 biomedical 도메인에 적용하기에 중요한 사항입니다. 이렇게 학습된 BioBERT 를 사용해서 NER, RE 그리고 QA 테스크에 적용했을 때, 기존의 SOTA 모델들 보다 좋은 성능을 달성합니다.
이상으로 BERT의 응용분야에 대한 논문을 알아보았어요. 이 논문을 읽다 보니 다양한 방면으로 transformer architecture가 사용될 수 있다는데 관심이 가네요. 다음 NLP 논문은 아마 BART나 T5 논문 리뷰가 될 것 같아요. 그럼 다음에 봐요~ 모두 안녕~
Reference
https://arxiv.org/abs/1901.08746
BioBERT: a pre-trained biomedical language representation model for biomedical text mining
Biomedical text mining is becoming increasingly important as the number of biomedical documents rapidly grows. With the progress in natural language processing (NLP), extracting valuable information from biomedical literature has gained popularity among re
arxiv.org



